DeepMind, el laboratorio londinense de inteligencia artificial que Google compró en 2014, ya ha creado programas que ganan siempre al ajedrez, al shogi y al Go, los juegos de tablero más complejos. Pero el objetivo final de la empresa no está en los pasatiempos, sino en resolver acuciantes problemas científicos. Su algoritmo AlphaFold, presentado a principios de diciembre en Cancún, México, ha ganado una competición mundial en predecir la estructura tridimensional de proteínas.
Predecir la estructura de cualquier proteína a partir de su secuencia de aminoácidos se considera uno de los santos griales de la biología. No es tarea menor; los aminoácidos son 20 moléculas con propiedades químicas ligeramente distintas, con lo cual están unidos por enlaces de diferentes longitudes y ángulos. Se tardaría más que la edad del universo en doblar una proteína por todas sus configuraciones posibles antes de dar con la estructura tridimensional correcta por azar.
Pese a lo esotérico de este campo científico, es difícil exagerar su importancia. Ciertas enfermedades, como el alzhéimer, el párkinson, la diabetes o la fibrosis quística, se deben a la acumulación de proteínas mal plegadas, algo que se podría evitar conocida la relación entre su secuencia y su estructura. Casi todos los medicamentos actúan por acoplamiento a la región específica de una proteína, un proceso que de nuevo depende de la estructura precisa de la diana. Además, con la capacidad de predecir exactamente cómo se doblará una cadena de aminoácidos, los científicos pueden diseñar proteínas artificiales, por ejemplo para degradar plásticos o compuestos contaminantes, del organismo o del medio ambiente.
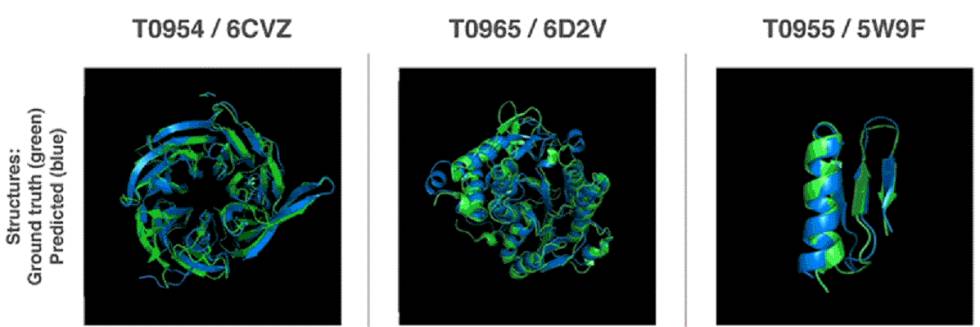
El equipo de Google, que se presentaba por primera vez al certamen, quedó primero de 98 concursantes, al estimar con mayor precisión la estructura de 25 de las 43 proteínas, según informa The Guardian. Para cada secuencia de aminoácidos, suele existir un pliegue correcto, que se corresponde a la configuración de mayor estabilidad bioquímica. En un laboratorio, se puede observar la forma real de las biomoléculas mediante técnicas como la resonancia magnética o la cristalografía de rayos X, un método parecido al que permitió a Rosalind Franklin ver por primera vez la estructura de la doble hélice del ADN.
La inteligencia artificial constituye un avance increíble con respecto a esas técnicas complejas y costosas, aunque todavía no es capaz de sustituirlas por completo. DeepMind entrenó a una red neuronal relacionando la forma y la secuencia génica de miles de proteínas conocidas. Armado con ese conocimiento, el programa AlphaFold predice la distancia y el ángulo entre cada pareja de aminoácidos de la cadena, y luego hace pequeños ajustes a la estructura completa para encontrar la configuración más estable.
No hay comentarios:
Publicar un comentario